BioMCP: Protocolo de contexto del modelo biomédico
BioMCP es un conjunto de herramientas de código abierto (licencia MIT) que dota a los asistentes y agentes de IA de conocimiento biomédico especializado. Desarrollado según el Protocolo de Contexto de Modelo (MCP), conecta los sistemas de IA con fuentes de datos biomédicos fiables, lo que les permite responder preguntas sobre ensayos clínicos, literatura científica y variantes genómicas con precisión y profundidad.
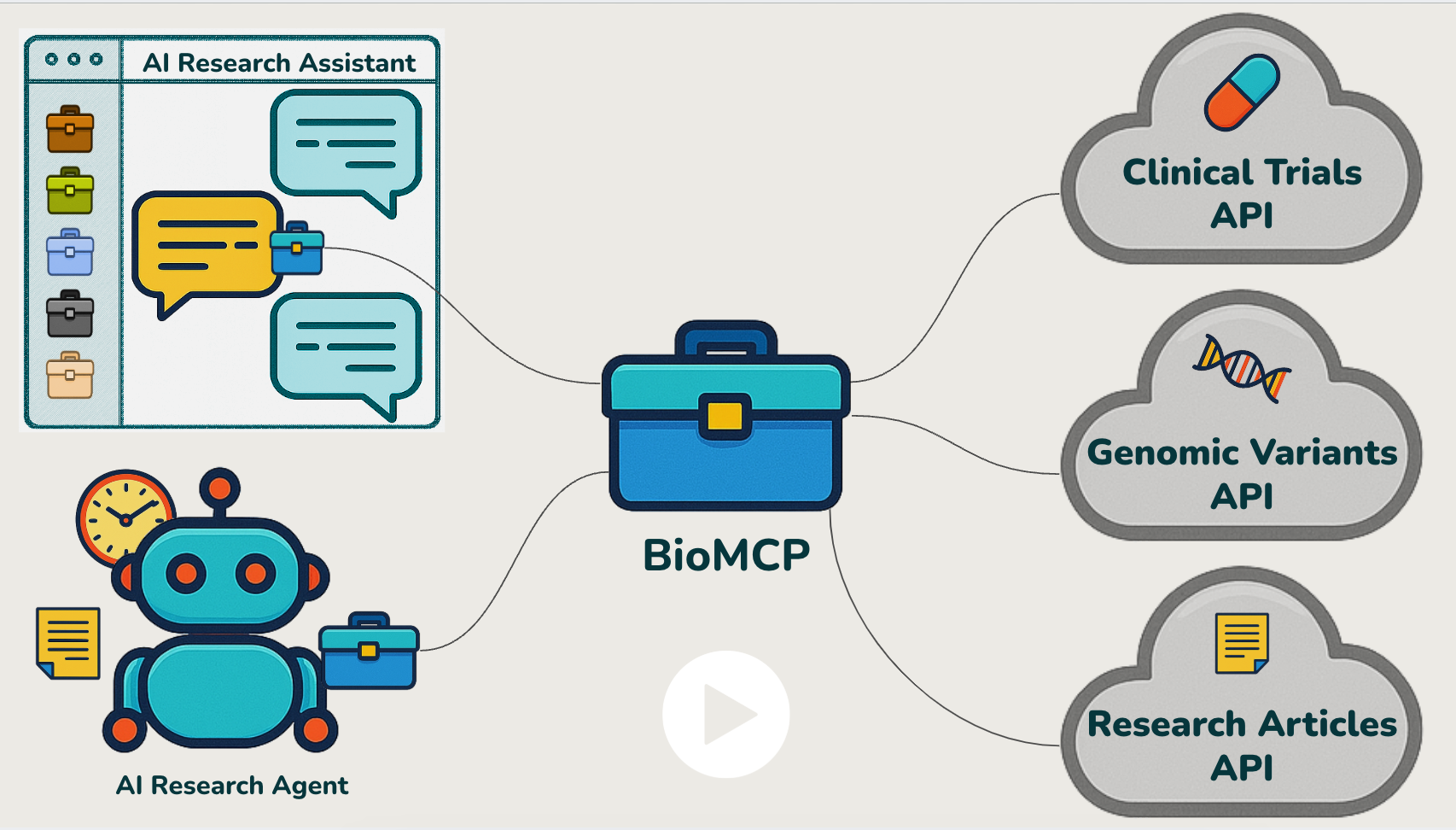
¿Por qué BioMCP?
Si bien los Modelos de Lenguaje Grande poseen un amplio conocimiento general, a menudo carecen de información especializada específica del dominio o de acceso a recursos actualizados. BioMCP cubre esta brecha en la biomedicina mediante:
Proporcionar acceso estructurado a ensayos clínicos, literatura biomédica y variantes genómicas.
Permitir consultas en lenguaje natural a bases de datos especializadas sin necesidad de conocer su sintaxis específica
Apoyar los flujos de trabajo de investigación biomédica a través de una interfaz consistente
Funciona como un servidor MCP para asistentes y agentes de IA
Related MCP server: Healthcare MCP Server
Fuentes de datos biomédicos
BioMCP se integra con tres fuentes de datos biomédicos clave:
PubTator3/PubMed : Literatura biomédica con anotaciones de entidades
ClinicalTrials.gov - Registro de ensayos clínicos y base de datos de resultados
MyVariant.info - Anotación consolidada de variantes genéticas de múltiples bases de datos
Herramientas MCP disponibles
PubMed y PubTator3
article_searcher: busca artículos por genes, enfermedades, variantes o palabras clavearticle_details: Obtenga información detallada del artículo, incluidos resúmenes y textos completos
ClinicalTrials.gov
trial_searcher: Búsqueda avanzada de ensayos con filtrado por condición, intervención, fase, etc.trial_protocol: Información detallada del protocolo del ensayotrial_locations: Ubicaciones de los sitios de prueba e información de contactotrial_outcomes: Resultados y medidas de resultadostrial_references: Publicaciones relacionadas
MiVariante.info
variant_searcher: busca variantes genéticas con filtrado sofisticadovariant_details: Anotaciones completas de múltiples fuentes (CIViC, ClinVar, COSMIC, dbSNP, etc.)
Inicio rápido
Para usuarios de Claude Desktop
Instalar si no lo tienes (recomendado):
# MacOS brew install uv # Windows/Linux pip install uvConfigurar Claude Desktop :
Abrir la configuración de Claude Desktop
Vaya a la sección Desarrollador
Haga clic en "Editar configuración" y agregue: GXP2
¡Reinicia Claude Desktop y comienza a chatear sobre temas biomédicos!
Instalación de paquetes de Python
Interfaz de línea de comandos
BioMCP proporciona una CLI integral para la interacción directa con la base de datos:
Pruebas y verificación
Pruebe su configuración BioMCP con el Inspector MCP:
Esto abre una interfaz web donde puedes explorar y probar todas las herramientas disponibles.
Versión empresarial: OncoMCP
OncoMCP amplía BioMCP con la plataforma de oncología de precisión de nivel empresarial (POP) de GenomOncology, que proporciona:
Implementación conforme a HIPAA : opciones seguras en las instalaciones
Coincidencia de pruebas en tiempo real : estado actualizado y coincidencia a nivel de brazo
Integración de la atención médica : Conectividad fluida entre EHR y almacén de datos
Base de conocimientos seleccionada : más de 15 000 ensayos y aprobaciones de la FDA
Emparejamiento sofisticado de pacientes : uso de perfiles clínicos y moleculares integrados
PNL avanzada : Extracción estructurada de texto no estructurado
Procesamiento integral de biomarcadores : procesamiento de mutaciones y reglas
Más información: GenomOncology
Registros MCP
Documentación
Para obtener documentación completa, visite https://biomcp.org
Informe de ejemplos de BioMCP
¿Quieres ver BioMCP en acción?
Consulta el repositorio complementario: 👉 biomcp-examples
Contiene indicaciones reales, informes de investigación generados por IA y evaluaciones con diferentes modelos. Úselo para explorar capacidades, comparar resultados o evaluar su propia configuración.
¿Tienes un ejemplo genial? ¡Nos encantaría que contribuyeras! Simplemente bifurca el repositorio y envía un PR con tu experimento.
Licencia
Este proyecto está licenciado bajo la licencia MIT.