Биомарт МКП
Сервер MCP для взаимодействия с Biomart
Model Context Protocol (MCP) — открытый протокол, который стандартизирует, как приложения предоставляют контекст LLM, разработанный Anthropic . Здесь мы используем MCP python-sdk для создания сервера MCP, который взаимодействует с Biomart через пакет pybiomart .
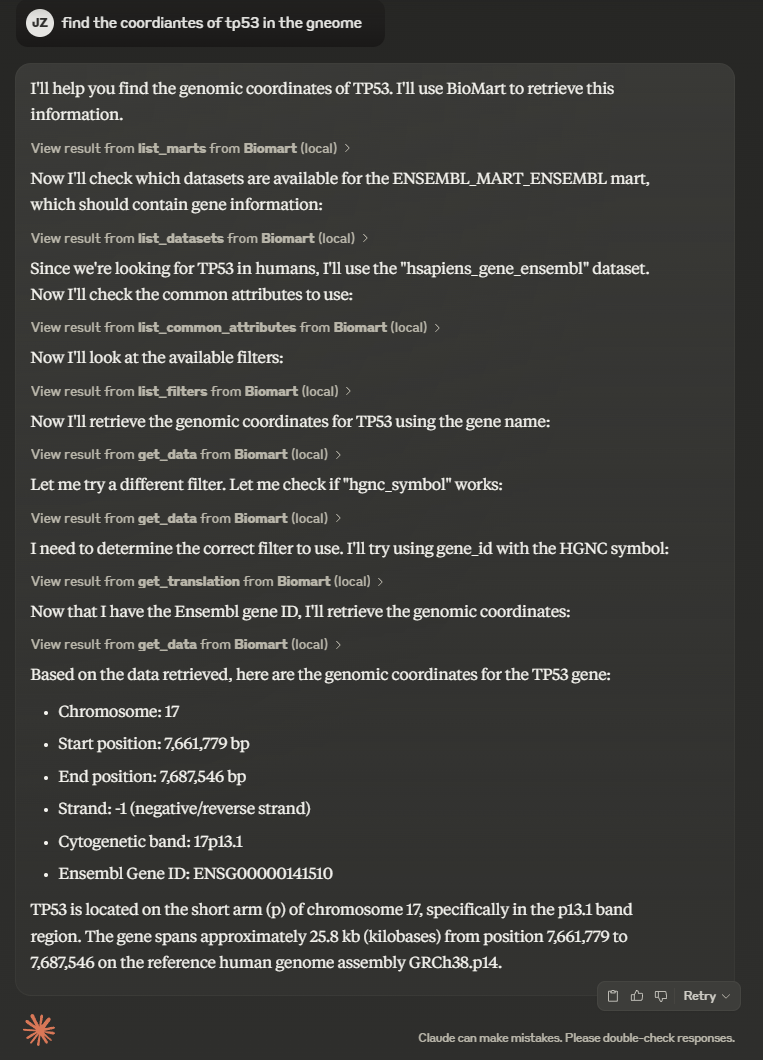
Имеется короткое демонстрационное видео, демонстрирующее работу сервера MCP на Claude Desktop.
Установка
Установка через Smithery
Чтобы автоматически установить Biomart MCP для Claude Desktop через Smithery :
Клонировать репозиторий
Клод Десктоп
Курсор
С помощью агентского режима Cusror другие модели также могут использовать преимущества серверов MCP, например, от OpenAI или DeepSeek. Щелкните шестеренку настройки курсора и перейдите в MCP , а затем либо добавьте сервер MCP в глобальную конфигурацию, либо добавьте его в область проекта, добавив .cursor/mcp.json в проект.
Пример .cursor/mcp.json :
Глама
Разработка
Related MCP server: MCP TapData Server
Функции
Biomart-MCP предоставляет несколько инструментов для взаимодействия с базами данных Biomart:
Поиск витрин и наборов данных : список доступных витрин и наборов данных для изучения структуры базы данных Biomart.
Исследование атрибутов и фильтров : просмотр общих или всех доступных атрибутов и фильтров для определенных наборов данных.
Извлечение данных : запросите Biomart с определенными атрибутами и фильтрами для получения биологических данных
Перевод идентификаторов : преобразование между различными биологическими идентификаторами (например, символы генов в идентификаторы ансамбля)
Внося вклад
Pull requests приветствуются! Несколько небольших заметок по разработке:
Мы намеренно используем здесь только
@mcp.tool(), чтобы обеспечить максимальную совместимость с клиентами, поддерживающими MCP, как показано в документации .Мы используем
@lru_cacheдля кэширования результатов функций, требующих больших вычислительных затрат или выполняющих внешние вызовы API.Нам нужно быть внимательными, чтобы не раздуть контекстное окно модели, например, вы увидите
df.to_csv(index=False).replace("\r", "")во многих местах. Этот возврат в стиле csv гораздо более эффективен с точки зрения токенов, чем что-то вродеdf.to_string(), где большинство токенов — это пробелы. Также помните о том, что извлечение всех генов из хромосомы или подобный большой запрос также будет слишком большим для контекстного окна.
Потенциальные будущие возможности
Конечно, есть еще много функций, которые можно добавить, некоторые из них, возможно, выходят за рамки имени biomart-mcp . Вот несколько идей:
Добавьте веб-скрапинг для сайтов ресурсов с
bs4, например, мы получили идентификатор гена Ensembl для NOTCH1, тогда, возможно, в некоторых случаях будет полезно извлечь сопоставленныеComments and Description Text from UniProtKBс его страницы на UCSC.$...$